4. Reakcja enzymatyczna
4.1. Równanie Michaelisa–Menten
W jednym z prostszych modeli reakcji enzymatycznych pojawia się równanie Michaelisa–Menten, ustanawiające zależność szybkość reakcji ![]() od stężenia substratu
od stężenia substratu ![]() :
:
| (4.1) |
Współczynniki ![]() i
i ![]() są pewnymi parametrami (
są pewnymi parametrami (![]() nazywa się stałą Michaelisa) — por. także wykład z Modeli matematycznych w biologii i medycynie. Wartości
nazywa się stałą Michaelisa) — por. także wykład z Modeli matematycznych w biologii i medycynie. Wartości ![]() i
i ![]() można zmierzyć doświadczalnie, [32] (zob. także [13]) podaje m.in. następujący zestaw danych:
można zmierzyć doświadczalnie, [32] (zob. także [13]) podaje m.in. następujący zestaw danych:
| 0.25 | 0.30 | 0.40 | 0.50 | 0.70 | 1.00 | 1.40 | 2.00 | |
| 2.4 | 2.6 | 4.2 | 3.8 | 6.2 | 7.4 | 10.2 | 11.4 |
Naszym zadaniem jest wyznaczenie takich wartości parametrów ![]() i
i ![]() , które najlepiej pasowałyby do powyższych danych eksperymentalnych.
, które najlepiej pasowałyby do powyższych danych eksperymentalnych.
4.1.1. Nietrafione uproszczenia
Wielu badaczy, jeszcze w czasach gdy obliczenia były zaporowo drogie, zaproponowało rozmaite transformacje powyższego problemu, pozwalające w ostatecznym rozrachunku sformułować zadanie jako liniowe zadanie najmniejszych kwadratów. Mając w pamięci numeryczną maksymę, że
Jeśli zadanie jest za trudne, należy… zmienić zadanie!
możemy ulec wrażeniu, że będzie to właściwa droga ku efektywnemu znalezieniu ,,optymalnych” wartości szukanych parametrów.
W przypadku zadania dopasowania ![]() i
i ![]() , używano (por. [10]) między innymi takich (matematycznie równoważnych) transformacji (4.1):
, używano (por. [10]) między innymi takich (matematycznie równoważnych) transformacji (4.1):
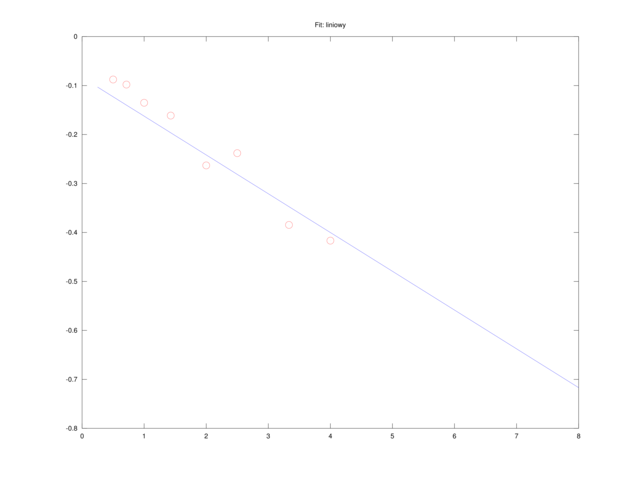
Równanie czysto liniowe
Mnożąc (4.1) stronami przez ![]() i dzieląc przez
i dzieląc przez ![]() , dostajemy równanie liniowe względem
, dostajemy równanie liniowe względem ![]() i
i ![]() :
:
| (4.2) |
które możemy oczywiście rozwiązać metodą najmniejszych kwadratów. Jeśli wektory kolumnowe S i v0 będą zawierać dane z pomiarów (odpowiednio: ![]() i
i ![]() ,
, ![]() ), to instrukcje
), to instrukcje
e = ones(size(S),1); A = [1./S, -1./v0]; b = -e; x = A \ b; V = x(2); Km = x(1);spowodują wyznaczenie
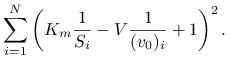 |
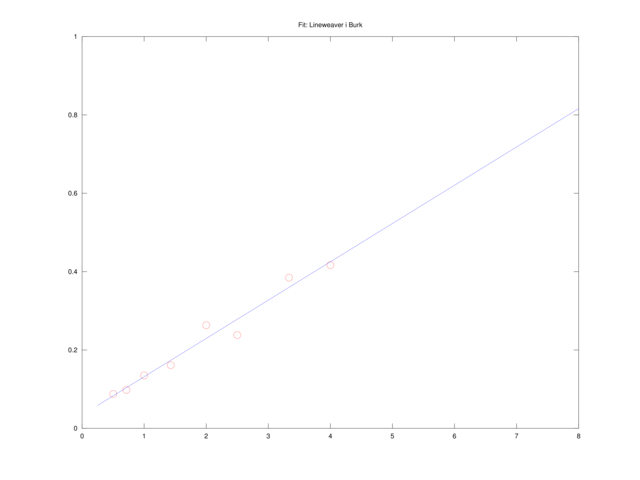
Równanie Lineweavera i Burka
Jeśli poprzednio wyprowadzone równanie podzielimy przez ![]() , dostaniemy równanie Lineweavera i Burka,
, dostaniemy równanie Lineweavera i Burka,
które jest liniowe względem pomocniczych zmiennych ![]() ,
, ![]() .
.
Analogicznie jak poprzednio, możemy znaleźć wartości ![]() , które zminimalizują wyrażenie
, które zminimalizują wyrażenie
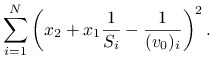 |
Rozwiążemy je w Octave, korzystając z operatora ,,\”, sekwencją komend:
A = [1./S, e]; b = 1./v0; x = A \ b; V = 1/x(2); Km = x(1)*V;
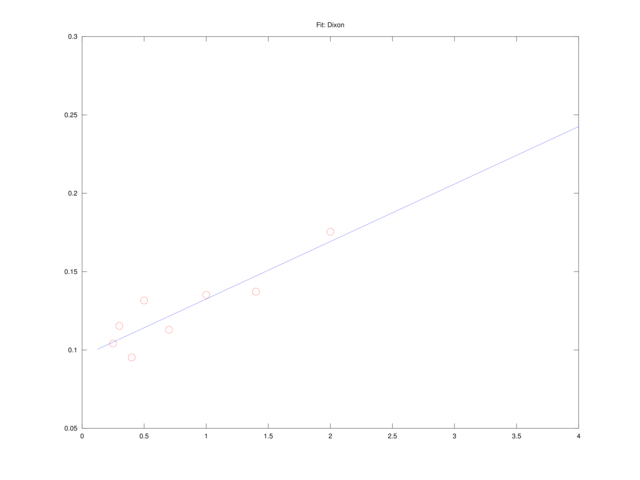
Równanie Dixona
Mnożąc równanie Lineweavera i Burka przez ![]() , dostajemy równanie Dixona,
, dostajemy równanie Dixona,
liniowe w pomocniczych zmiennych ![]() ,
, ![]() . Ponownie więc możemy dopasować
. Ponownie więc możemy dopasować ![]() i
i ![]() , rozwiązując liniowe zadanie najmniejszych kwadratów.
, rozwiązując liniowe zadanie najmniejszych kwadratów.
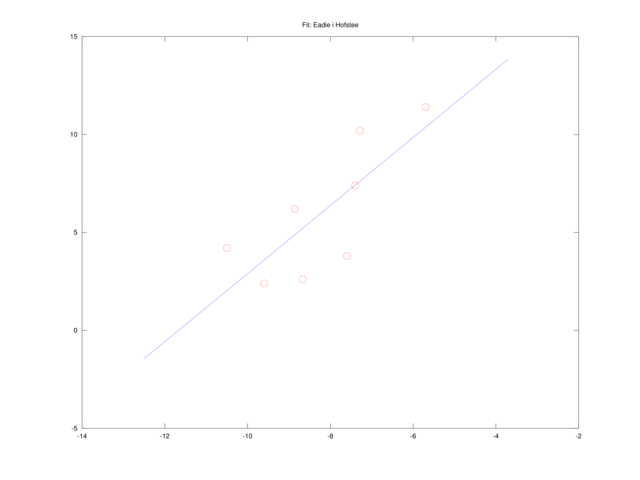
Równanie Eadie i Hofstee
Mnożąc wreszcie równanie liniowe (4.2) przez ![]() , dochodzimy do równania postaci:
, dochodzimy do równania postaci:
które jest liniowe w oryginalnych zmiennych, ![]() i
i ![]() . To zadanie też możemy rozwiązać przez liniowe zadanie najmniejszych kwadratów.
. To zadanie też możemy rozwiązać przez liniowe zadanie najmniejszych kwadratów.
Ćwiczenie 4.1
Dla danych podanych w tabeli na początku rozdziału, wyznacz każdym z opisanych sposobów współczynniki ![]() i
i ![]() .
.
Przykładowe rozwiązania znajdują się w listingu w dalszej części wykładu. Swoją odpowiedź możesz porównać z tabelką w następnym rozdziale.
4.1.1.1. Krytyka metod prowadzących do liniowego zadania najmniejszych kwadratów
Powstaje więc — bynajmniej nie filozoficzne, ale czysto praktyczne — pytanie, która z metod jest ,,lepsza”, czyli która z nich da najlepsze możliwe dopasowanie (przy rozsądnym koszcie). Otóż wszystko zależy od tego, co będziemy rozumieli pod pojęciem ,,najlepsze”: wszak z definicji każde z uzyskanych przez nas rozwiązań było najlepsze, jako rozwiązanie zadania minimalizacji residuum postaci ![]() dla zadanych
dla zadanych ![]() i
i ![]() .
.
Ponieważ ,,naturalnym” sformułowaniem naszego zadania było (4.1), wydaje się równie naturalnym postawienie i ocena rozwiązania w sensie nieliniowego zadania najmniejszych kwadratów:
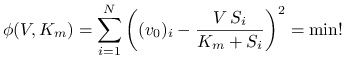 |
(4.3) |
Jeśli porównać wartości ![]() dla parametrów otrzymanych wyżej opisanymi metodami, to okaże się, że
dla parametrów otrzymanych wyżej opisanymi metodami, to okaże się, że
| równanie | uwarunkowanie ( |
|||
| Lineweaver i Burk | 29.43 | 2.88 | 1.71 | 12.1 |
| Dixon | 27.27 | 2.61 | 1.58 | 7.13 |
| Eadie i Hofstee | 20.27 | 1.74 | 2.65 | 874 |
| liniowe | 12.00 | 0.95 | 23.40 | 3170 |
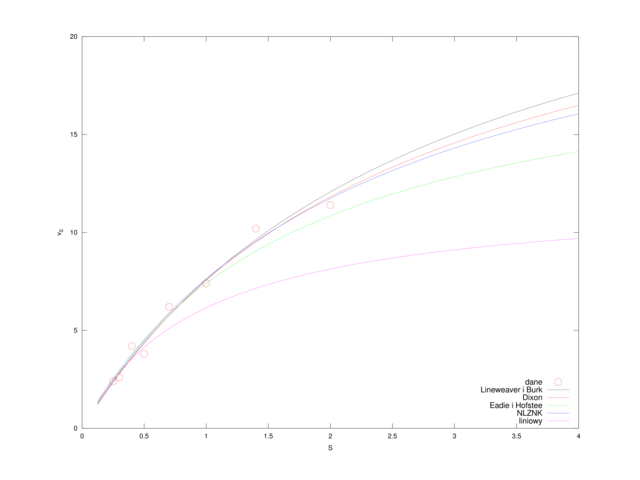
A więc w tym sensie, zdecydowanie najlepsze rezultaty dało dopasowanie metodą Dixona (por. także rysunek 4.5). Co więcej, okazuje się, że metody prowadzące do liniowej zależności od ![]() i
i ![]() , czyli równanie liniowe (4.2) i równanie Eadie i Hofstee są bardzo źle uwarunkowane10Współczynnik uwarunkowania zadania odzwierciedla podatność wyniku na zaburzenia danych. Gdy zadanie jest źle uwarunkowane (tzn. ma bardzo duży współczynnik uwarunkowania), nawet małe zaburzenie danych może spowodować bardzo duży błąd wyniku., ze współczynnikiem uwarunkowania
, czyli równanie liniowe (4.2) i równanie Eadie i Hofstee są bardzo źle uwarunkowane10Współczynnik uwarunkowania zadania odzwierciedla podatność wyniku na zaburzenia danych. Gdy zadanie jest źle uwarunkowane (tzn. ma bardzo duży współczynnik uwarunkowania), nawet małe zaburzenie danych może spowodować bardzo duży błąd wyniku., ze współczynnikiem uwarunkowania ![]() rzędu
rzędu ![]() . Jest to o tyle ważne, że dane do naszego zadania zdają się być zmierzone z dokładnością względną
. Jest to o tyle ważne, że dane do naszego zadania zdają się być zmierzone z dokładnością względną ![]() rzędu
rzędu ![]() : taki błąd może więc skutkować zaburzeniem względnym wyniku na poziomie rzędu
: taki błąd może więc skutkować zaburzeniem względnym wyniku na poziomie rzędu ![]() , czyli na poziomie
, czyli na poziomie ![]() .
.
Sensowne wyniki dają metody Lineweaver i Burk oraz Dixona, ale… czy nie można jeszcze lepiej? Wszak możemy spróbować rozwiązać wprost nieliniowe zadanie najmniejszych kwadratów (4.3), na przykład korzystając z solvera sqp, dostępnego w Octave.
4.1.2. Rozwiązanie nieliniowego zadania najmniejszych kwadratów
Solversqp jest narzędziem służącym do rozwiązywania znacznie bardziej złożonych zadań optymalizacji, dlatego jego użycie bywa skomplikowane. Ale w naszym prostym przypadku — minimalizacji bez ograniczneń, dla funkcjonału kwadratowego — nie będziemy musieli podawać mu zbyt wielu parametrów. Najpierw jednak zdefiniujemy funkcjonał, który będziemy minimalizować:
% (X(1), X(2)) <---> (V, K) F = @(X,S) (X(1)*S)./(X(2)+S); phi = @(X) sumsq(v0 - F(X,S));
Jak widać, zaczęliśmy od określenia funkcji
która pojawia się w definicji (4.1) i w (4.3), i która z pewnością nam się przyda: na przykład do narysowania wykresu ![]() dla wyznaczonych
dla wyznaczonych ![]() i
i ![]() (dlatego definiujemy ją od razu wektorowo ze względu na
(dlatego definiujemy ją od razu wektorowo ze względu na ![]() ). Potem, przy użyciu
). Potem, przy użyciu ![]() , zdefiniowaliśmy
, zdefiniowaliśmy ![]() , przy czym — ponieważ
, przy czym — ponieważ phi jest anonimową funkcją tylko jednego argumentu, X, to w jej definicji zostaną użyte wcześniej przez nas zdefiniowane wektory S i v0, zawierające dane zadania. Dzięki temu, łatwo nam będzie wyznaczyć sumę kwadratów współrzędnych wektora v0 - F(X,S) — do tego właśnie służy funkcja Octave sumsq.
Teraz wystarczy wywołać solver sqp na naszym funkcjonale ![]() , z początkowym przybliżeniem
, z początkowym przybliżeniem ![]() wyznaczonym np. metodą Dixona:
wyznaczonym np. metodą Dixona:
% podane na wejściu sqp parametry (V, Km) zostały wyznaczone metodą Dixona [x, phix, info, iter, nphi] = sqp([V;Km], phi) V = x(1); Km = x(2);
W wyniku dostajemy ![]() oraz
oraz ![]() , dla których
, dla których ![]() , a więc (nieco) lepiej niż dotychczas! Jak zwykle w przypadku metod iteracyjnych, zażądamy możliwie wielu informacji diagnostycznych, takich jak: kod zakończenia
, a więc (nieco) lepiej niż dotychczas! Jak zwykle w przypadku metod iteracyjnych, zażądamy możliwie wielu informacji diagnostycznych, takich jak: kod zakończenia info, liczba wykonanych iteracji iter, oraz wywołań funkcjonału nphi. Pożądana wartość info to, zgodnie z manualem, 101: zakończenie z powodu nikłego postępu iteracji. Musimy jednak pamiętać, że znalezione minimum może być jedynie minimum lokalnym i wybór innego punktu startowego może dać w rezultacie znacznie lepszy (lub gorszy) wynik.
Podsumowując, w dzisiejszych czasach, gdy nawet dość zaawansowae obliczenia są (w miarę) łatwe i tanie, nie powinniśmy bać się nieliniowości. Pamiętajmy także, że jeśli tylko możemy sensownie wspomóc się przybliżeniem uzyskanym na drodze rozsądnej linearyzacji — warto z tej opcji skorzystać.
Na marginesie dodajmy, że opisane przez nas wcześniej metody sprowadzenia zadania do zadania liniowego, są wciąż rutynowo stosowane w innych zadaniach dopasowywania do punktów pomiarowych wykresu, na przykład postaci ![]() : biorąc logarytm od obu stron, ponownie dostajemy zadanie liniowe na
: biorąc logarytm od obu stron, ponownie dostajemy zadanie liniowe na ![]() i
i ![]() :
: ![]() . Jednak, jak już doświadczyliśmy, transformacja zadania najczęściej powoduje też zmianę sposobu, w jaki mierzymy błąd dopasowania. Niektóre transformacje dodatkowo mogą wyolbrzymiać lub redukować różnice pomiędzy danymi punktami pomiarowymi.
. Jednak, jak już doświadczyliśmy, transformacja zadania najczęściej powoduje też zmianę sposobu, w jaki mierzymy błąd dopasowania. Niektóre transformacje dodatkowo mogą wyolbrzymiać lub redukować różnice pomiędzy danymi punktami pomiarowymi.
S = [0.25 ; 0.30; 0.40; 0.50; 0.70; 1.00; 1.40; 2.00];
v0 = [2.4; 2.6; 4.2; 3.8; 6.2; 7.4; 10.2; 11.4];
F = @(X,S) (X(1)*S)./(X(2)+S);
fitfun = @(X) sumsq(v0 - F(X,S));
function cbA = lsqcond(A, b, x, tol)
if nargin < 4
tol = 1e2;
end
c = norm(A*x)/norm(b);
t = norm(A*x-b)/norm(A*x);
condA = cond(A);
cb = condA/c;
cA = (condA^2)*t + condA;
cbA = cb+cA;
if (cbA) > tol
warning(['Uwarunkowanie zadania: ', num2str([cb, cA])]);
end
end
p = 0; n = 100;
s = linspace(0.125,4,n);
e = ones(size(S));
model = 5;
A = [1./S, -1./v0]; b = -e;
x = A \ b;
V(model) = x(2); Km(model) = x(1);
resid(model) = fitfun([V(model),Km(model)]);
cbA(model) = lsqcond(A,b,x);
plot(A(:,1), A(:,2), 'ro'); hold on;
plot(1./s, -(1+x(1)./s)/x(2),'-b'); hold off;
title('Fit: liniowy'); pause(p);
print('-depsc','linear.eps');
% Lineweaver i Burk
model = 1;
A = [1./S, e]; b = 1./v0;
x = A \ b;
V(model) = 1/x(2); Km(model) = x(1)*V(model);
resid(model) = fitfun([V(model),Km(model)]);
cbA(model) = lsqcond(A,b,x);
plot(A(:,1), b, 'ro'); hold on;
plot(1./s, x(1)./s+x(2),'-b'); hold off;
title('Fit: Lineweaver i Burk'); pause(p);
print('-depsc','lib.eps');
% Dixon
model = 2;
A = [e, S]; b = S./v0;
x = A \ b;
V(model) = 1/x(2); Km(model) = x(1)*V(model);
resid(model) = fitfun([V(model),Km(model)]);
cbA(model) = lsqcond(A,b,x);
plot(A(:,2), b, 'ro'); hold on;
plot(s, x(2)*s+x(1),'-b'); hold off;
title('Fit: Dixon'); pause(p);
print('-depsc','dixon.eps');
% Eadie i Hofstee
model = 3;
A = [-v0./S, e]; b = v0;
x = A \ b;
V(model) = x(2); Km(model) = x(1);
resid(model) = fitfun([V(model),Km(model)]);
cbA(model) = lsqcond(A,b,x);
plot(A(:,1), b, 'ro'); hold on;
s3 = linspace(min(A(:,1))-2, max(A(:,1))+2, 100);
plot(s3, x(1)*s3+x(2),'-b'); hold off;
title('Fit: Eadie i Hofstee'); pause(p);
print('-depsc','eih.eps');
% nonlinear fit; X = (V,Km)
model = 4;
[x, fitfunx, info, iter, nF, lambda] = sqp([V(2);Km(2)], fitfun);
V(model) = x(1); Km(model) = x(2);
resid(model) = fitfun([V(model),Km(model)]);
cbA(model) = NA;
if info ~= 101
warning('SQP: problemy z rozwiazaniem zadania');
end
printf('+------+---------+--------+----------+----------+\n');
printf(' Model | V | K | resid | cond\n');
printf('+------+---------+--------+----------+----------+\n');
printf(' %2d | %4.2f | %4.2f | %6.2f | %4.2e\n', [1:length(V);V;Km;resid;cbA]);
printf('+------+---------+--------+----------+----------+\n');
plotname = {'dane','Lineweaver i Burk','Dixon','Eadie i Hofstee','NLZNK','liniowy'};
plot(S, v0, 'ro');
hold on;
for i = 1:length(V)
plot(s, F([V(i),Km(i)],s) , ['-', num2str(i-1)]);
pause(p);
end
hold off;
xlabel('S'); ylabel('v_0');
legend(plotname, 'location', 'southeast');
print('-depsc','michaelis-fit.eps')
4.2. Różniczkowy model łańcucha reakcji enzymatycznych
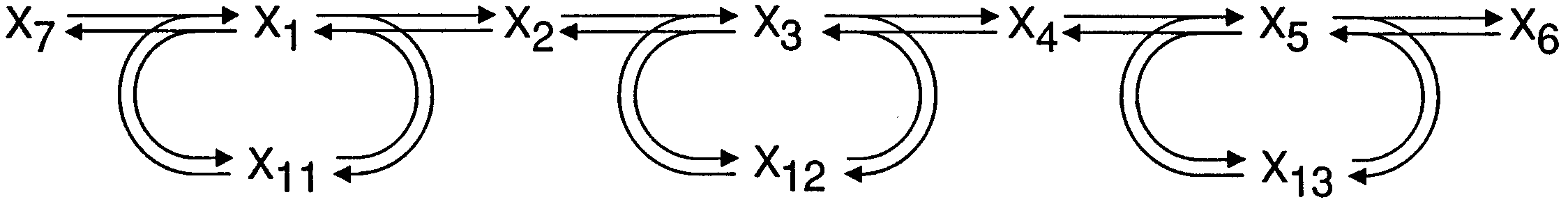
W rozdziale 6. pracy [24] rozważa się model łańcucha trzech reakcji katalizowanych enzymatycznie. Niech ![]() oznacza (dane) stężenie substratu,
oznacza (dane) stężenie substratu, ![]() i
i ![]() — (niewiadome) stężenie dwóch produktów pośrednich, a
— (niewiadome) stężenie dwóch produktów pośrednich, a ![]() — (dane) stężenie produktu finalnego reakcji. Dalej, niech
— (dane) stężenie produktu finalnego reakcji. Dalej, niech ![]() będą zadanymi całkowitymi stężeniami trzech enzymów katalizujących każdą z reakcji (zob. rysunek 4.6 pochodzący z (Źródło: [24, rysunek 9]), przy czym
będą zadanymi całkowitymi stężeniami trzech enzymów katalizujących każdą z reakcji (zob. rysunek 4.6 pochodzący z (Źródło: [24, rysunek 9]), przy czym ![]() będą nieznanymi stężeniami odpowiednich enzymów związanych z produktami pośrednimi (a przez to nieczynnymi). Stężenia wolnych enzymów oznaczymy przez
będą nieznanymi stężeniami odpowiednich enzymów związanych z produktami pośrednimi (a przez to nieczynnymi). Stężenia wolnych enzymów oznaczymy przez ![]() ,
, ![]() ,
, ![]() .
.
Wtedy kinetyka powyższego układu może być modelowana następującym zestawem równań różniczkowych zwyczajnych [24, równania (75)—(82)]11Podajemy go już po pewnych uproszczeniach.:
z parametrami ![]() ,
uzupełnionego warunkiem początkowym dla
,
uzupełnionego warunkiem początkowym dla ![]() . Naszym zadaniem jest wyznaczenie wykresu zależności prędkości (netto)
. Naszym zadaniem jest wyznaczenie wykresu zależności prędkości (netto) ![]() powstawania produktu od czasu
powstawania produktu od czasu ![]() , zgodnie ze wzorem [24, równanie (83)]
, zgodnie ze wzorem [24, równanie (83)]
W tym celu w rutynowy sposób wyznaczymy rozwiązanie powyższego układu równań, korzystając z funkcji lsode. Najpierw jednak zapiszemy układ równań w formie takiej, by występowały w nim jedynie niewiadome ![]() oraz zadane parametry
oraz zadane parametry ![]() :
:
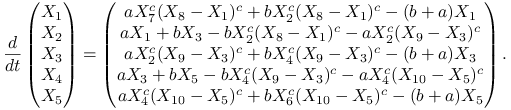 |
W naszych eksperymentach numerycznych przyjmiemy za [24] ![]() ,
, ![]() ,
, ![]() , a także
, a także ![]() . Ponadto
. Ponadto ![]() (badając model tak, jak w [24], sprawdzalibyśmy wpływ zmiany tego parametru na zmianę
(badając model tak, jak w [24], sprawdzalibyśmy wpływ zmiany tego parametru na zmianę ![]() ),
), ![]() . W chwili
. W chwili ![]() , ustalamy
, ustalamy ![]() . Oczywiście, cały skrypt symulujący przebieg reakcji zapiszemy w formie sparametryzowanej tak, by móc łatwo zmieniać wartości wszystkich danych zadania.
. Oczywiście, cały skrypt symulujący przebieg reakcji zapiszemy w formie sparametryzowanej tak, by móc łatwo zmieniać wartości wszystkich danych zadania.
Funkcja prawej strony miałaby więc postać12Jeżeli w definicji dX przypadkowo napiszesz dX = [ a -b; ..itd..], to interpreter Octave zrozumie, że chodzi o macierz o dwóch kolumnach, dX = [ a, -b; ..itd..]. Dlatego, powinniśmy tutaj konsekwentnie pisać dX = [ a - b; ..itd..] (z operatorem odejmowania otoczonym spacjami).
function dX = reaction(X,t) global a; global b; global c; global X6; global X7; global X8; global X9; global X10; dX = [a*(X7*(X8-X(1)))^c + b*(X(2)*(X8-X(1)))^c - (b+a)*X(1) ; a*X(1) + b*X(3) - b*(X(2)*(X8-X(1)))^c - a*(X(2)*(X9-X(3)))^c ; a*(X(2)*(X9-X(3)))^c + b*(X(4)*(X9-X(3)))^c - (b+a)*X(3) ; a*X(3) + b*X(5) - b*(X(4)*(X9-X(3)))^c - a*(X(4)*(X10-X(5)))^c ; a*(X(4)*(X10-X(5)))^c + b*(X6*(X10-X(5)))^c - (b+a)*X(5)]; end(dla większej skuteczności iloczyny postaci
global a; global b; global c; global X6; global X7; global X8; global X9; global X10; a = 2; b = 1; c = 4; X6 = X8 = X9 = X10 = 1; X7 = 1.425; T = 10; N = 800; t = linspace(0,T,N); X = lsode(@reaction, zeros(5,1), t); plot(t,X); pause(3); v = a*X(:,5) - b*(X6*(X10-X(:,5))).^c; plot(t,v)
Tradycyjnie, aby nieco upewnić się co do jakości otrzymanych wyników, powinniśmy przeprowadzić kilka symulacji z różnymi parametrami tolerancji błędu (i zbadać, czy przypadkiem nie padliśmy ofiarą aliasingu, czyli zbyt małej rozdzielczości wizualizacji, fałszującej rzeczywisty przebieg rozwiązania).
Ćwiczenie 4.2
Przeprowadź testy wizualnej i numerycznej jakości uzyskanego rozwiązania, zmieniając N (by zabezpieczyć się przed aliasingiem) oraz lsode_options (by zmniejszyć ryzyko wzięcia numerycznych artefaktów za prawdziwe własności rozwiązania).